regression_multiple
index
Multiple Regression
Beispiel: Everitt (2010) car cleaning data
Daten einlesen, erster grafischer Eindruck
# Daten einlesen
car.data <- read.delim("http://www.psych.uni-goettingen.de/de/mat/mv/everitt-car-data.txt")
# variablen direkt in namespace holen
attach(car.data)
# einen Eindruck der Daten bekommen (erste 10 Zeilen)
car.data[1:10,]
# eine Korrelationsmatrix
cor(car.data)
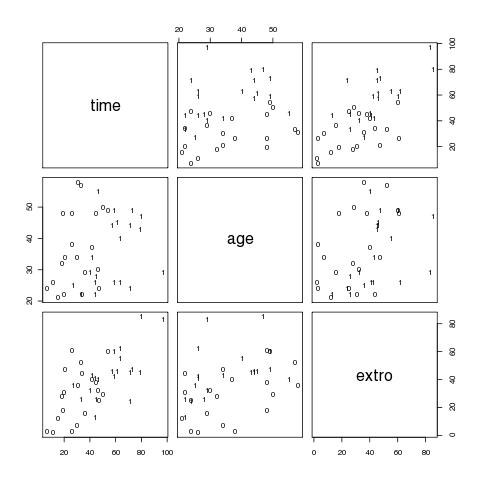
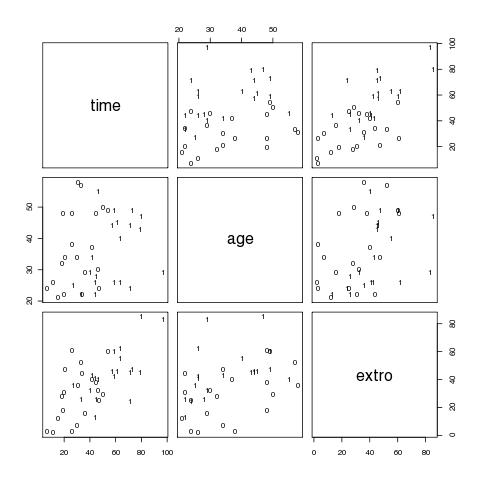
# ein erster grafischer Überblick (Fig 4.1)
# pairs erzeugt multiple scatterplots
# panel legt den Inhalt der einzelnen Fenster fest
# text trägt die Werte in der Spalte sex an die jeweils geplotteten x-y-Koordinaten aus den übergebenen Matrix-Spalten
# die 0 und 1 zeigen die Lage der verschiedenen AVen für Frauen bzw Männer
pairs(cbind(time,age,extro),panel=function(x,y) text(x,y,sex))
Linear Model spezifizieren, Anpassung rechnen lassen
# mit den drei Prädiktoren sex, age und extro
# Kurzergebnis ausgeben lassen
summary(lm(time~sex+age+extro))
# Summary zwei Prädiktoren sex, extro
summary(lm(time~sex+extro))
# ergibt output
Call:
lm(formula = time ~ sex + extro)
Residuals:
Min 1Q Median 3Q Max
-26.193 -9.474 -2.149 10.165 23.918
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 15.6797 4.4365 3.534 0.001118 **
sex 19.1801 4.4727 4.288 0.000124 ***
extro 0.5093 0.1151 4.423 8.24e-05 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 12.95 on 37 degrees of freedom
Multiple R-squared: 0.632, Adjusted R-squared: 0.6121
F-statistic: 31.77 on 2 and 37 DF, p-value: 9.284e-09
# Omnibus F-Test (F-statistic) prüft Hyp., dass alle Regressionskoeffizienten gleich 0 sind.
# bzw. ob durch die Vorhersage-Linearkombination ein signifikanter Anteil der Varianz am Kriterium erklärt wird.
# Multiple R-squared: Anteil der Varianz des Kriteriums, der durch die Kombination der Prädiktoren gebunden/erklärt wird.
# Adjusted R-squared: rechnet R-squared so um, dass die Anzahl der erklärenden Terme im Modell berücksichtigt wird.
# adjusted R-squared steigt im Gegensatz zu R-squared nur, wenn der neue Term das Modell um mehr als durch Zufall erwartet verbessert.
# adjusted R-squared kann negativ sein und ist immer <= R-squared.

p Anzahl der Regressore im linear Model (ohne constant), n ist sample-size.
# unter Coefficients finden sich die unstandardisierten Beta-Gewichte mit ihrem Standardfehler sowie ein T-Test zur Prüfung des Einzeleffekts # (t-Werte sind Estimate / Std. Error)
Vorhersage eines einzelnen Kriteriumswertes (Vp 3) mit Prädiktor sex und extero (zu Fuß)
# lm-Objekt speichern m.lm <- lm(time~sex+extro) # Summary-Objekt speichern s.lm <- summary(m.lm) # was gibt es im Summary-Objekt? names(s.lm) # nur die Koeffizienten brauchen wir koeff <- s.lm$coefficients[,1] # Vp 3 Vorhersagewert (fitted value) berechnen e.time.3 <- koeff['(Intercept)'] + koeff['sex'] * sex[3] + koeff['extro'] * extro[3] e.time.3 # Vorhersagewerte finden sich im lm-Objekt unter fitted.values m.lm$fitted.values # müßte gleich sein dem 3. Wert in den Vorhersagewerten m.lm$fitted.values[3]
Vorhergesagte Werte mit predict
predict(m.lm)
Standardisierte Beta-Gewichte sind vergleichbarer
# Standardisierte Beta-Gewichte: # Vars standardisieren car.data['st.sex'] <- car.data$sex / sd(car.data$sex) car.data['st.age'] <- car.data$age / sd(car.data$age) car.data['st.extro'] <- car.data$extro / sd(car.data$extro) car.data['st.time'] <- car.data$time / sd(car.data$time) # vergleichende Summaries # unstandardisiert summary(lm(car.data$time~car.data$sex+car.data$extro)) # Prädiktoren standardisiert summary(lm(car.data$time~car.data$st.sex+car.data$st.extro)) # Kriterium und Prädiktoren standardisiert summary(lm(car.data$st.time~car.data$st.sex+car.data$st.extro)) # Alternative: scale() benutzen und die Standardisierng 'on the fly' machen anstelle eine neue Variable zu bilden summary(lm(scale(car.data$time) ~ scale(car.data$sex) + scale(car.data$extro))) # Alternative: standardisierte Regressionskoeffizienten aus den unstandardisieren errechnen # standardisierte Regressionskoeffizienten lassen sich auch einfach ausrechnen # raw-coeffizient(predictor) * sd(predictor) / sd(criterion) # das unstandardisierte Modell speichern ureg <- lm(car.data$time~car.data$sex+car.data$extro) # die standardisierten Regressionskoeffizienten berechnen und ausgeben ureg$coefficients['car.data$sex'] * sd(car.data$sex) / sd(car.data$time) ureg$coefficients['car.data$extro'] * sd(car.data$extro) / sd(car.data$time)
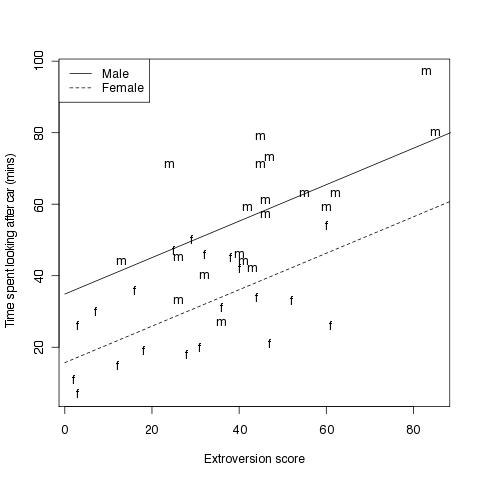
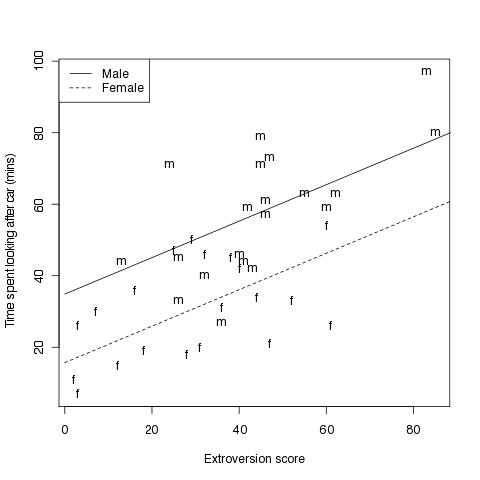
Einen Scatterplot erstellen mit den beiden Regressionsgeraden für das jeweilige Geschlecht
#Fig 4.2
# vergleichend die beiden Regressionsgeraden für Männer und Frauen
# Grafikumgebung zurücksetzen
par(mfrow=c(1,1))
# einen Vektor mit 'm' für Maänner und 'f' für Frauen erstellen
Sex<-rep("m",length(sex))
Sex[sex==0]<-"f"
# einen Vektor mit 50 Elementen erstellen die in regelmäßigen Abständen zwischen 0 und 90 liegen
x<-seq(0,90,length=50)
# die Geradengleichungen mit derselben Steigung und verschiedenem Y-Achsenabschnitt
ym<-15.68+19.18+0.51*x
yf<-15.68+0.51*x
# und nun der plot
plot(car.data$time ~ car.data$extro, # plotte Grundmodell
xlab="Extroversion score", # X-Achsen-Beschriftung
ylab="Time spent looking after car (mins)",type="n") # Y-Achsen-Beschriftung
text(car.data$extro,car.data$time,labels=Sex) # an die Koordinaten den Text (Buchstaben) ausgeben
lines(x,ym,lty=1) # Regressionsgerade für Männer einzeichnen, Linienart festlegen
lines(x,yf,lty=2) # dito für Frauen
legend("topleft",c("Male","Female"), # eine Legende in die linke, obere Ecke
lty=1:2) # zugeordneter Linientyp in der Legende, der dem der Regressionsgeraden entspricht
Wechselwirkungsterm mit in MR einbeziehen
# Daten vorbereiten
time <- car.data$time
sex <- car.data$sex
extro <- car.data$extro
# das Modell anpassen und Summary ausgeben
rr <- lm(time~sex+extro+sex:extro)
summary(rr)
# ergibt output
Call:
lm(formula = time ~ sex + extro + sex:extro)
Residuals:
Min 1Q Median 3Q Max
-24.807 -9.445 -1.677 10.477 27.183
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 20.0182 5.4560 3.669 0.000782 ***
sex 7.8178 9.5705 0.817 0.419379
extro 0.3607 0.1590 2.268 0.029430 *
sex:extro 0.3052 0.2279 1.339 0.188970
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 12.81 on 36 degrees of freedom
Multiple R-squared: 0.6495, Adjusted R-squared: 0.6203
F-statistic: 22.23 on 3 and 36 DF, p-value: 2.548e-08
# Alternativ kann dasselbe Modell angepasst werden,
# indem ein neuer Prädiktor aus dem Produkt der alten berechnet und in das Modell mit eingefügt wird
mult.sex.extro <- sex * extro
rr2 <- lm(time ~ sex + extro + mult.sex.extro)
summary(rr2)
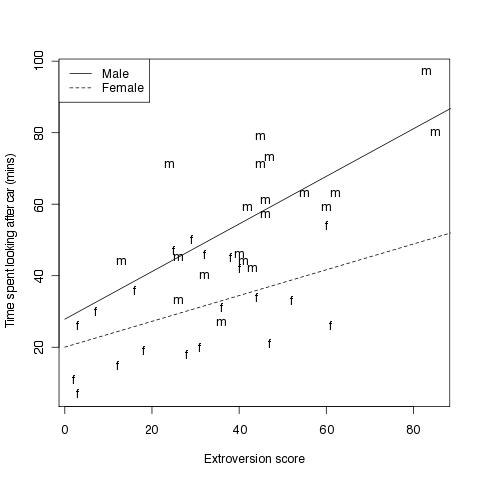
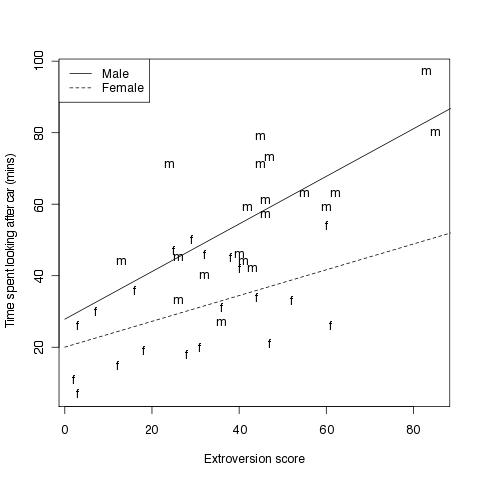
#Fig 4.3
# Abbildung die zusätzlich noch die Wechselwirkung mit einbezieht
# Grafik vorbereiten
Sex<-rep("m",length(sex))
Sex[sex==0]<-"f"
x<-seq(0,90,length=50)
# für Männer ist x = 1
#ym<-20+7.82+(0.36+0.31)*x
# eine Gerade über den Wertebereich von x mit 1
ym <- rr$coefficients['(Intercept)'] + rr$coefficients['sex'] * 1 + (rr$coefficients['extro'] * 1 + rr$coefficients['sex:extro'] * 1) * x
# für Frauen ist x = 0
# yf<-20+0.36*x
yf <- rr$coefficients['(Intercept)'] + rr$coefficients['sex'] * 0 + (rr$coefficients['extro'] * 1 + rr$coefficients['sex:extro'] * 0) * x
plot(time~extro,xlab="Extroversion score",
ylab="Time spent looking after car (mins)",type="n")
text(extro,time,labels=Sex)
lines(x,ym,lty=1)
lines(x,yf,lty=2)
legend("topleft",c("Male","Female"),lty=1:2)
Prüfung auf Multikollinearität und Prädiktorenselektion mit Course-Data
# Daten einlesen
course.data <- read.delim("http://www.psych.uni-goettingen.de/de/mat/mv/course-data.txt")
# und die Spalten in den Namespace holen
attach(course.data)
# ein Einblick in die Struktur des Data.Frames
course.data[1:10,]
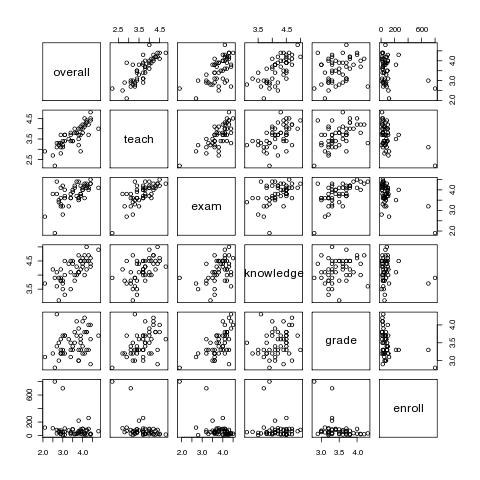
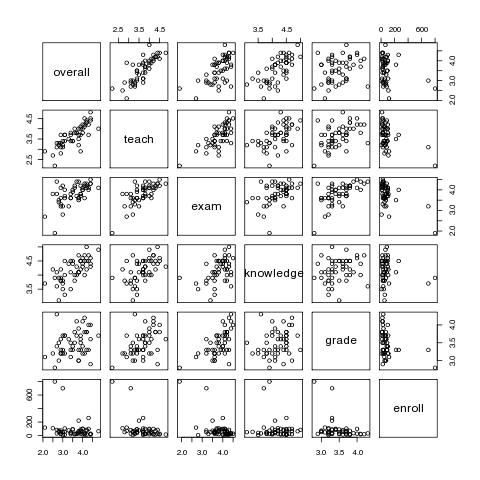
# ein erster grafischer Überblick
pairs(course.data)
# oder
plot(course.data)
Kontrolle auf Probleme mit Multikollinearität mit dem VIF (Variance Inflation Factor). VIF wird gebildet aus der multiplen Korrelation eines Prädiktors mit dem Rest der Prädiktoren. Daumenregel: Wenn der VIF 10 überschreitet gibt es Probleme mit Kollinearität im Pool der Prädiktoren.
Der VIF kann leicht errechnet werden, indem man die Restprädiktoren in ein 'lm' steckt als Prädiktoren und den relevanten Prädiktor als Kriterium. Das multiple R^2 finet man im Summary.
Die Formel für VIF ist 1 / (1 - R^2)
vif.teach <- 1 /(1 - summary(lm(teach ~ exam + knowledge + grade + enroll))$r.squared) vif.teach vif.exam <- 1 /(1 - summary(lm(exam ~ teach + knowledge + grade + enroll))$r.squared) vif.exam vif.knowledge <- 1 /(1 - summary(lm(knowledge ~ teach + exam + grade + enroll))$r.squared) vif.knowledge vif.grade <- 1 /(1 - summary(lm(grade ~ teach + exam + knowledge + enroll))$r.squared) vif.grade vif.enroll <- 1 /(1 - summary(lm(enroll ~ teach + exam + knowledge + grade ))$r.squared) vif.enroll
Jede Änderung an den Prädiktoren bzw. den Vpn verändert das Gesamtgefüge und erfordert eine Neuanpassung des Modells. Das gilt bei der Herausnahme oder beim Hinzufügen eines einzigen Prädiktors oder natürlich einer Gruppe. Entscheidungen über das weitere Vorgehen müssen bei jedem Einzelschritt durch Neuanpassung überprüft werden.
forward: Anfangen mit dem Prädiktor mit der höchsten Einzelkorrelation, dann je den vielversprechendsten Prädiktor neu mit aufnehmen
backward: Anfangen mit allen Prädiktoren und dann den je 'schwächsten' aus dem Modell nehmen.
stepwise: Kombination: Schrittweise hinzunehmen und dann prüfen, ob aus den bereits integrierten Variablen sich eine als nicht mehr bedeutsam erweist.
AIC: Akaike's information criterion.
AIC 'bestraft' die Hinzunahme von Variablen, rechnet aber deren zusätzlichen Erklärungswert gegen. Frage, die das AIC beantwortet: Wie ist der Verlust an Erklärung im Vergleich mit dem Gewinn durch ein sparsameres Modell?
Kriterium: AIC soll kleiner werden (auch im Minus-Bereich)
# Backward regression # volles Modell generieren reg<-lm(overall~teach+exam+knosuwledge+grade+enroll) # backward step(reg, direction="backward") # Kriterium: AIC soll kleiner werden (auch im Minus-Bereich) # 'step' kann auch 'forward' und 'both'
Beurteilung einzelner Fälle, standardized residuals, deletion-residuals, leverage und Cook's-distance
Leverage
lever <- hatvalues(m.lm)
Cook's distance: Einfluss eines Falles auf die Parameterschätzung des Gesamtmodells. Ein Wert größer 1 heisst, dass diese Beobachtung einen unverhältnismäßig großen Einfluss hat (Empfehlung Everitt, 2010).
Die Funktion cooks.distance(), der das lineare Modell übergeben wird, liefert die cook's distance für die Beobachtungen zurück.
Ein plot() des Modells liefert mehrere Grafiken u. a. verschiedene Residual-Plots sowie einen Plot der die Cook's Distance visualisiert