index
Einfache Regression
Ein paar Basics
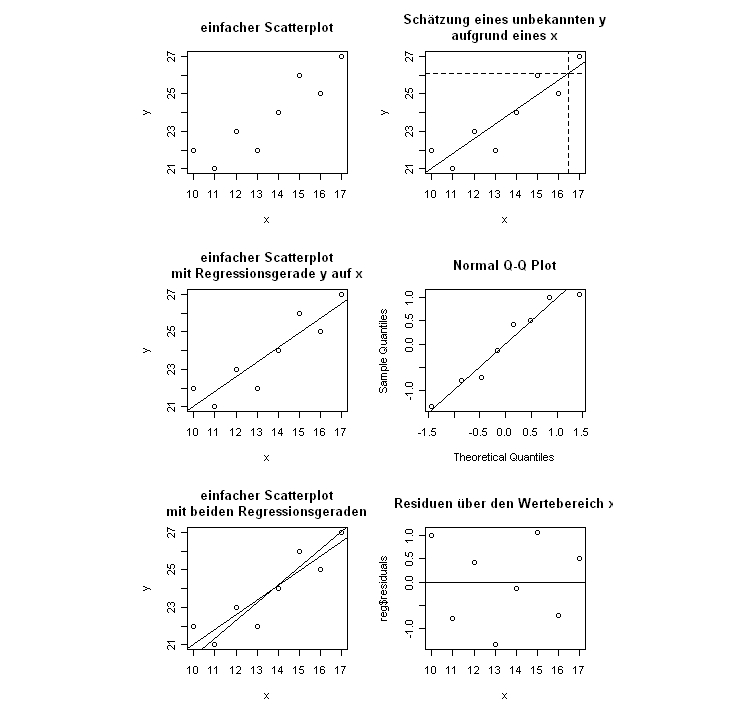
# einfache Regression # Produkt Moment Korrelation x <- c(10,11,12,13,14,15,16,17) y <- c(22,21,23,22,24,26,25,27) cor(x,y) # Visualisierung Scatterplot x <- c(10,11,12,13,14,15,16,17, 15, 17, 10) y <- c(22,21,23,22,24,26,25,27, 22, 22, 15) plot(x, y) # oder mit Nullpunkt (veränderte Achsen) plot(x, y, ylim=c(-5,30), xlim=c(-5,20)) abline(h=0) abline(v=0) #? Regression: Vorhersage der y bei gegebenen x model <- lm(y ~ x) # die Koeffizienten (Y-Achsen-Abschnitt, Steigung) model # einzeichnen abline(model) # Zweite Regressionsgerade einzeichnen # Modell Rechnen ymodel <- lm(x ~ y) # Koeffizienten ymodel # Die Koeffizienten wären nur direkt einsetzbar, wenn man die x- und y-Achsen austauschen würde. # Durch Umrechnen kann man sie in den bestehenden Plot einfügen. # Constant (y-Achsenabschnitt): Bezogen auf die bestehende Grafik gibt y.c den x-Wert an, bei dem y = 0 # Ansatz: y = y.s * x + y.c # für y = 0 # 0 = y.s * x + y.c # auflösen nach x # 0 - y.c = y.s * x # (0 - y.c)/ y.s = x # da in aktueller Grafik y und x vertauscht sind, ist dies der y-Achsenabschnitt y.c <- ymodel$coefficients[1] # Steigung: 1 / y.s # y.s sind x-Einheiten pro 1 y für den bestehenden Plot daher Umrechnung nötig. y.s <- ymodel$coefficients[2] print(c(y.c, y.s)) abline((0 - y.c) / y.s, 1/y.s, lty=4) # alles was ich über oben gerechnete Regression erfahren kann attributes(model) # alternativ nur die Namen der Attribute des 'Auswertungs-Objektes' names(model) # Zugriff über objekt['attribut-name'] model['fitted.values'] # vorhergesagte Werte model$fitted.values # alternativer Zugriff # z. B. die Residuen residuals(model) # alternativ natürlich model$residuals # oder die Parameter für die Geradengleichung coefficients(model) # par 1 ist Prädiktor-Achsenabschnitt, par 2 ist Steigung(Multiplikator für Kriterium) # Abbildungen machen # sechs vergleichende Abbildungen neben- bzw übereinander vorbereiten: nf<-layout(matrix(c(1,4,2,5,3,6),3,2,byrow=TRUE),c(1,1),c(1,1),TRUE) # Siehe /r/graph/multiple reg <- lm(y ~ x) plot(y ~ x, main='einfacher Scatterplot') plot(y ~ x, main='einfacher Scatterplot\nmit Regressionsgerade y auf x') abline(reg) plot(y ~ x, main='einfacher Scatterplot\nmit beiden Regressionsgeraden') abline(reg) ylm <- lm(x ~ y) yc <- coefficients(ylm) abline((0 - yc[1])/yc[2], 2 - yc[2]) # Schätzung für ein x = 16.5 plot(y ~ x, main='Schätzung eines unbekannten y\naufgrund eines x') abline(reg) abline(v=16.5, lty=2) # den Schätzwert von y einzeichnen abline(h=reg$coefficients[1] + reg$coefficients[2] * 16.5, lty=2) # Normalverteilung der Residuen qqnorm(reg$residuals) # wenn man möchte kann man auch die Winkelhalbierende als Gerade durchlegen abline(c(0,0), c(1,1)) # alternativ abline(0,1) # Plot der Residuen gegen die Prädiktoren plot(x, reg$residuals, main='Residuen über den Wertebereich x') abline(h=0) # Plot der Residuen gegen die vorhergesagten Werte (predicted values) #plot(reg$fitted.values, reg$residuals, main='Residuen über predicted values x')
Ein Prädiktor
Plot machen, Regressionsgerade fitten, einzeichnen, 95% Konfidenzintervall anzeigen, Modellparameter ansehen.
# Daten einlesen
dlm <- read.delim("http://www.psych.uni-goettingen.de/de/mat/mv/virtual-lm.txt")
# Grafikumgebung reset
par(mfrow=c(1,1))
# zwei vergleichende Abbildungen übereinander vorbereiten:
nf<-layout(matrix(c(1,2,4,3),2,2,byrow=TRUE),c(1,1),c(1,1),TRUE)
# der Standardweg
# d2 durch d1 vorhersagen
# Modell rechnen und das Ergebnisobjekt speichern
reg <- lm(dlm$d2 ~ dlm$d1)
# ein erster Plot. Achtung: Im Plot-Befehl wird auch das Modell angegeben
plot(dlm$d2 ~dlm$d1)
# das hat den Vorteil, dass auch die Regressionsgerade einzeichnen dem Modell entnommen werden kann
abline(reg)
# Sind die Residuen normalverteilt? grafische Überprüfung.
qqnorm(reg$residuals,main="NV der Residuen?")
# Residuen ansehen
plot(dlm$d1, reg$residuals, main="Residuen gegen Prädiktor")
# und die Null-Linie
abline(h=0)
# Residuen gegen vorhergesagte Y-Werte
#plot(reg$fitted.values, reg$residuals, main="Residuen gegen Prädiktor")
# oder äquivalent mit Model-Schreibweise
#plot(reg$residuals ~ reg$fitted.values, main="Residuen gegen Prädiktor2")
# Grundplot
plot(dlm$d2 ~dlm$d1)
abline(lm(dlm$d2 ~dlm$d1))
# Berechnung der Standardfehler der Schätzung
# predict errechnet vorhergesagte Werte für Modelle
# se.fit ist ein Flag, das die Streuung des Vorhersagefehlers mit berechnet
# alles wird im Auswertungsobjekt pred gespeichert
pred<-predict(reg, se.fit=TRUE)
fitval <- pred$fit
se <- pred$se.fit
# Vorbereitung für die Visualisierung der SE-Verläufe
# den Prädiktor in einen Datenvektor 'index' speichern, der die Positionen der d1-Werte enthält, wenn sie der Größe nach geordnet werden
index <- order(dlm$d1)
# die Werte müssen der Größe nach geordnet geplottet werden, damit die Linien nicht hin- und herspringen und ein Knäuel in der Abbildung entsteht.
y <- fitval[index] # die zugehörigen vorhergesagten Werte ermitteln
stde <- se[index] # die zugehörigen Standardfehler ermitteln
yu<-y+1.96*stde # einen Datenvektor mit den Obergrenzen des Konfidenzintervalls bauen
yl<-y-1.96*stde # dasselbe mit den Untergrenzen
# und nun die beiden Verläufe einzeichnen
lines(dlm$d1[index],yu,lty=2)
lines(dlm$d1[index],yl,lty=2)
Ein Prädiktor aber mehrere Komponenten davon
In ein Modell kann zusätzlich zu der linearen Komponente auch eine quadratische oder höhere Komponente ein- und desselben Prädiktors hinzugefügt werden.
# Daten einlesen
lm.data <- read.delim("http://www.psych.uni-goettingen.de/de/mat/mv/virtual-lm.dat")
# welche Spalten/Variablen gibt es und wie heissen sie?
names(lm.data)
# ein erster grafischer Überblick über die Verhältnisse
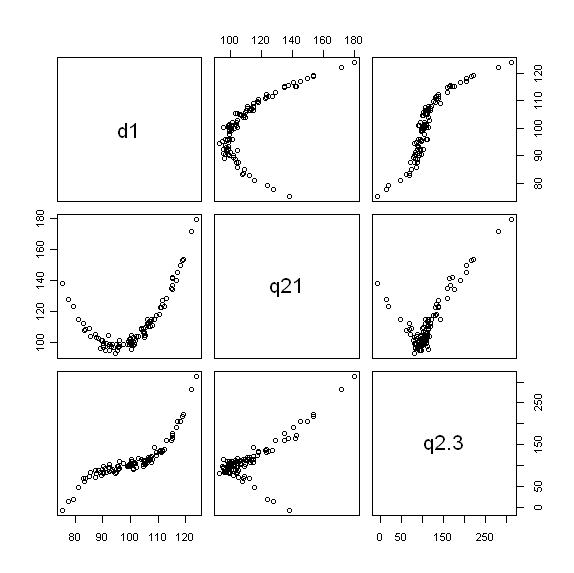
pairs(lm.data[c('d1','q21', 'q2.3')])
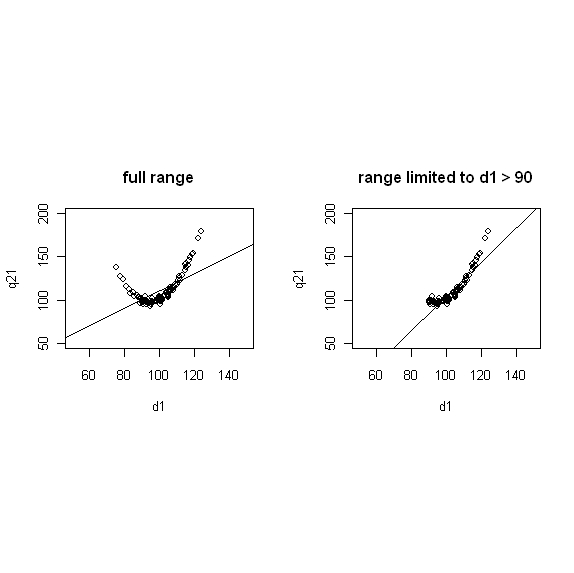
# zwei vergleichende Abbildungen übereinander vorbereiten: nf<-layout(matrix(c(1,2,0,0),2,2,byrow=TRUE),c(2,1),c(2,2),TRUE) # zwei vergleichende Abbildungen nebeneinander vorbereiten (vertikal gestreckt): par(mfrow = c(1, 2)) # zwei vergleichende Abbildungen nebeneinander vorbereiten: nf<-layout(matrix(c(1,2,0,0),2,2,byrow=TRUE),c(2,2),c(2,0),TRUE) # vier vergleichende Abbildungen neben- bzw übereinander vorbereiten: nf<-layout(matrix(c(1,2,0,0),2,2,byrow=TRUE),c(2,2),c(2,0),TRUE) # q21 über d1 vorhersagen # simple linear model r.l <- lm(lm.data$q21 ~lm.data$d1) summary(r.l) plot(lm.data$d1, lm.data$q21, ylim=c(50,200), xlim=c(50,150), xlab="d1", ylab="q21", main="full range") # und die Regressionsgerade dazu abline(lm(r.l)) # eine angepasste Linie kann täuschend gut sein # nur die d1 > 90 berücksichtigen xx <- lm.data$d1[lm.data$d1 > 90] yy <- lm.data$q21[lm.data$d1 > 90] rr <- lm(yy ~xx) summary(rr) plot(xx, yy, ylim=c(50,200), xlim=c(50,150), xlab="d1", ylab="q21", main="range limited to d1 > 90") # und die Regressionsgerade dazu abline(rr)
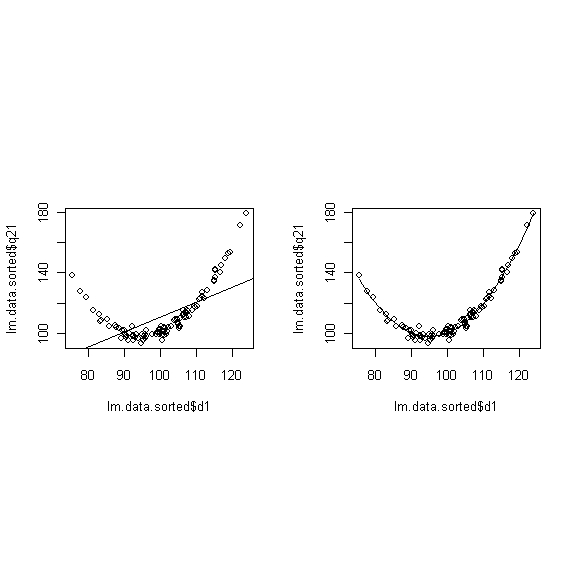
# eine quadratische Komponente von d1 mit dazu nehmen # zwei vergleichende Abbildungen nebeneinander vorbereiten: nf<-layout(matrix(c(1,2,0,0),2,2,byrow=TRUE),c(2,2),c(2,0),TRUE) # für eine vernünftige Darstellung muss die Tabelle nach d1 sortiert werden lm.data.sorted <- lm.data[order(lm.data$d1),] dq <- lm.data.sorted$d1^2 # Das Modell rechnen lassen r.l <- lm(lm.data.sorted$q21 ~lm.data.sorted$d1) r.lq <- lm(lm.data.sorted$q21 ~lm.data.sorted$d1 + dq) # Wie sehen die Parameter aus? summary(r.lq) # und zwei Abbildungen dazu # Abb 1, nur Linie als Modell plot(lm.data.sorted$q21 ~lm.data.sorted$d1) abline(lm(r.l)) # Abb2, quadratische Komponente hinzugenommen plot(lm.data.sorted$q21 ~lm.data.sorted$d1) lines(lm.data.sorted$d1,r.lq$fit)
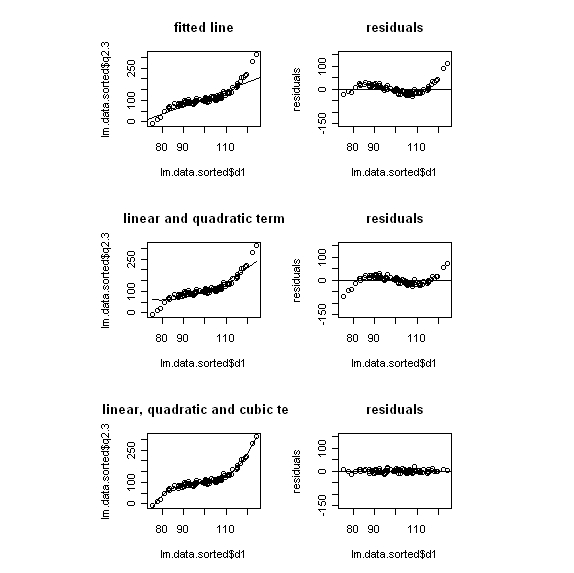
# eine lineare, eine quadratische und eine cubische Komponente von d1 mit dazu nehmen # vergleichende Abbildungen nebeneinander vorbereiten: nf<-layout(matrix(c(1,2,3,4,5,6),3,2,byrow=TRUE),c(1,1,1),c(1,1),TRUE) # für eine vernünftige Darstellung muss die Tabelle nach d1 sortiert werden lm.data.sorted <- lm.data[order(lm.data$d1),] dq <- lm.data.sorted$d1^2 dc <- lm.data.sorted$d1^3 # Das Modell rechnen lassen r.l <- lm(lm.data.sorted$q2.3 ~lm.data.sorted$d1) r.lq <- lm(lm.data.sorted$q2.3 ~lm.data.sorted$d1 + dq) r.lqc <- lm(lm.data.sorted$q2.3 ~lm.data.sorted$d1 +dq + dc) # Wie sehen die Parameter aus? summary(r.l) summary(r.lq) summary(r.lqc) # und Abbildungen dazu # Abb 1, nur Linie als Modell plot(lm.data.sorted$q2.3 ~ lm.data.sorted$d1, main="fitted line") abline(lm(r.l)) # Residuen dazu plot(r.l$residuals ~lm.data.sorted$d1, ylab="residuals", ylim=c(-150,150), main="residuals") abline(h=0) # Abb2, quadratische Komponente hinzugenommen plot(lm.data.sorted$q2.3 ~lm.data.sorted$d1, main="linear and quadratic term") lines(lm.data.sorted$d1,r.lq$fit) # Residuen dazu plot(r.lq$residuals ~lm.data.sorted$d1, ylab="residuals", ylim=c(-150,150), main="residuals") abline(h=0) # Abb3, cubische Komponente hinzugenommen plot(lm.data.sorted$q2.3 ~lm.data.sorted$d1, main="linear, quadratic and cubic term") lines(lm.data.sorted$d1,r.lqc$fit) # Residuen dazu plot(r.lqc$residuals ~lm.data.sorted$d1, ylab="residuals", ylim=c(-150,150), main="residuals") abline(h=0)
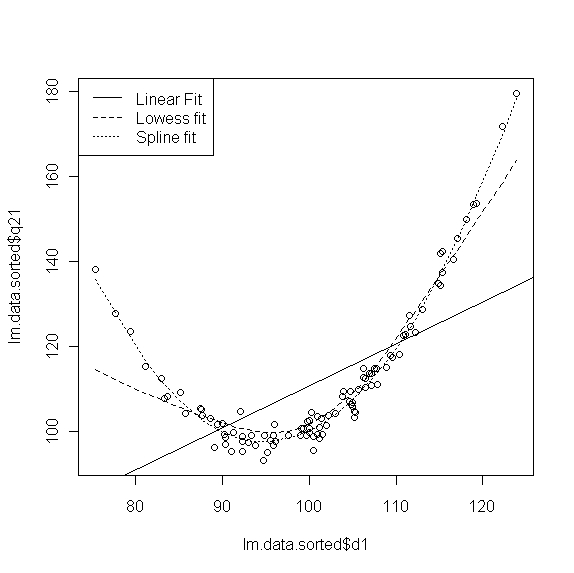
Ein Modell mit Hilfe von Smoothing-Techniken (Spline) anpassen.
Hier handelt es sich nicht um ein mathematisches Modell des Zusammenhanges von Prädiktor und Kriterium, sondern um eine grafische Techniken (Glättung). Hier kurz angerissen sind "lowess-fit" und "spline".
# Spline anpassen
# Grafikumgebung reset
par(mfrow=c(1,1))
plot(lm.data.sorted$q21 ~ lm.data.sorted$d1)
abline(lm(lm.data.sorted$q21 ~ lm.data.sorted$d1))
lines(lowess(lm.data.sorted$q21 ~ lm.data.sorted$d1),lty=2)
lines(smooth.spline(lm.data.sorted$d1, lm.data.sorted$q21),lty=3)
legend("topleft",c("Linear Fit","Lowess fit","Spline fit"),lty=1:3)
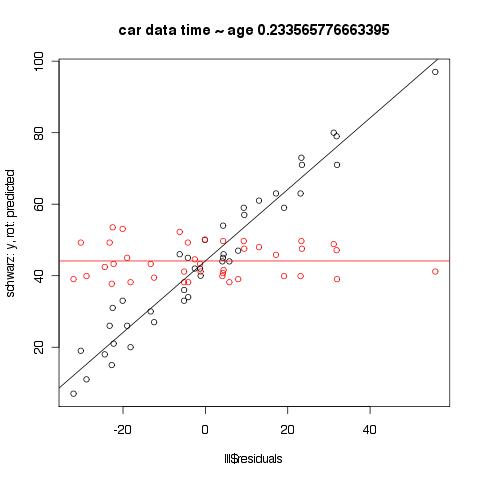
Zur Frage des Unterschiedes zwischen Residualplot geschätzte Werte vs reale Werte
Der Plot der Residuen gegen die geschätzten Werte ist geeigneter, um Hinweise auf Besonderheiten in den Residuen zu bekommen, die z. B. ein anderes Modell nahelegen könnten. Man kann im Plot unten schön den Effekt der Regression zur Mitte sehen, der bei den geschätzten Werten nicht vorhanden ist.
data <- read.delim("http://www.psych.uni-goettingen.de/de/mat/mv/everitt-car-data.txt")
lll <- lm(data$time ~ data$age)
plot(lll$residuals, data$time,
main = paste("car data time ~ age", cor(data$time, data$age), sep=" ", collapse = NULL),
ylab='schwarz: y, rot: predicted')
points(cbind(lll$residuals, lll$fitted.values), type='p', pch=21, col="red")
ll.res <- lm(lll$fitted.values ~ lll$residuals)
abline(ll.res, col="red")
ll.res <- lm(data$time ~ lll$residuals)
abline(ll.res)
-